목차
1. Abstract
2. Experiment
3. Result
4. Discussion
5. Reference
2. Experiment
3. Result
4. Discussion
5. Reference
본문내용
DNA를 만드는 단계이다. 이번 실험에서는 pfu polymerase를 통해 primer를 연장하여 새로운 DNA 가닥을 만든다. 원하는 PCR 산물의 크기가 크거나 반응효소의 농도가 낮을 때는 시간을 연장시킬 수도 있다.
(4) 전기영동 기기에 Agarose gel을 넣고 1X TAE buffer를 잠길 때까지 채운다.
(5) 1kbp DNA ladder와 6X Loading star를 섞어 Agarose gel의 첫 번째 홈에 loading 한다.
(6) PCR 샘플과 6X Loading star를 섞어 두 번째 홈에 loading 한다.
(7) 100 V의 전압에서 1시간 동안 전기영동을 실시한다.
(8) Agarose gel을 꺼내 UV transilluminator 위에 놓고 UV를 쬐어 PCR의 결과를 확인한다.
※ 실험 시 유의사항
- 의 온도조절에 유의한다. 가 너무 높으면 프라이머가 주형 DNA에 너무 약하게 결합되어 PCR산물의 양이 너무 적고, 가 너무 낮으면 프라이머가 비특이적으로 결합해 원하지 않는 DNA가 증폭될 수 있기 때문이다.
- 핵산 분해효소는 손이나 땀 등에도 있기 때문에 혼입을 막기 위해 장갑과 마스크를 반드시 착용해야 한다.
- 사용하는 tube와 피펫 팁, 용액 등은 멸균해 핵산 분해효소를 불활성 시켜둬야 한다.
3. Result
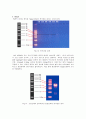
PCR의 결과로 증폭된 Target DNA의 전기영동 결과는 다음과 같다.
Fig 13. 전기영동 결과
이번 실험에서 모든 조의 전기영동 결과가 대체로 희미하게 나왔고, 1조와 4조와 6조가 조금 모양이 보이며 그 중 1조가 가장 잘 보이는 상태이다. 희미한 부분을 첫 번째 홈에 loading한 DNA ladder (기준이 되는 샘플)와 우리 6조의 전기영동 결과를 비교해보면, 2.0kbp과 3.0kbp 사이에 위치하며 3.0kbp쪽에 더 가까우므로 대략 2.8kb라고 측정할 수 있다. 하지만 결과가 매우 희미해 정확한 확인이 쉽지 않아 조교님께서 추가적으로 정상상태의 밴드사진을 올려주셨다. 이를 통해 정상상태의 Target DNA의 크기를 측정해보자.
Fig 14. 조교님께서 첨부해주신 정상상태의 전기영동 결과
Fig 14.를 살펴보면, 정상상태의 밴드 또한 이번 실험 결과와 마찬가지로 Target DNA의 크기가 대략 2.8kbp라는 것을 알 수 있다.
4. Discussion
전기영동 결과 우리 조의 밴드 중 가장 밝은 부분의 크기를 측정하니 대략 2.8kbp였다. 하지만 우리 조를 포함한 모든 조의 밴드가 대체적으로 너무 흐릿하고 희미하게 나와 정확한 판별이 힘들었다. 조교님이 첨부해주신 정상상태 밴드에서의 크기를 측정하니 마찬가지로 2.8kbp로 측정되었다. 이렇게 전기영동의 결과 Target DNA의 크기가 2.8kbp인 이유와 전기영동 결과가 희미하게 제대로 나오지 않은 이유에 대해 분석해보자.
1) 전기영동의 결과 Target DNA의 크기가 2.8kbp인 이유
이번 실험에 사용한 plasmid DNA는 유전자 재조합된 pET-22b(+)이며, PCR mixture 제작에 사용한 Forward primer는 T7 primer(T7 promoter), Reverse primer는 T7 Terminator primer이다. pET-22b(+)의 염기 서열 중 이 두 프라이머의 sequence landmarks는 다음과 같다.
Fig 15. T7 promoter와 T7 Terminator의 sequence landmarks
따라서 T7 promotor는 361-377, T7 terminator는 26-72이고, Forward primer와 Reverse primer는 T7 promoter와 T7 Terminator의 부위에 상보적으로 결합해 DNA의 합성을 진행하므로 우리가 기대할 수 있는 Target DNA의 크기는 (377-26= 351bp =0.351kbp)이다. 그러나 eGFP (0.8kpb)와 another gene (1.7kbp) 또한 함께 증폭되었을 것이다. 따라서 우리가 이번에 사용한 복제할 DNA의 크기인 2.5kbp에 0.351kbp의 길이를 더해주면 이번 실험에서 얻을 수 있는 이론적인 Target DNA의 크기는 2.851kbp가 나오게 된다.
2) 전기영동 결과가 희미하게 제대로 나오지 않은 이유
먼저 가장 큰 오차의 원인은 PCR mixture를 만드는 과정에서 발생했다고 생각한다. PCR mixture를 만들기 위해 넣은 시약 중 Reverse primer (T7 terminator primer)는 넣어야 하는 양이 0.5 μl로 극도로 적은 양이었기 때문에 가장 적은 용량의 마이크로 피펫과 피펫 팁을 사용해 정확히 넣어보려 했지만 거의 눈에 보이지 않을 정도로 작아 용액이 들어갔는지 확인하는 것조차 쉽지 않았다. 이러한 정확하게 넣지 못한 용액의 양으로 인해 오차가 발생했을 수 있다고 생각한다. Reverse primer는 Target DNA의 증폭에 있어 그 범위를 정해주는 매우 중요한 시약이기 때문에 정량의 오차가 PCR 과정에서의 오류를 유발시킬 수 있다. 또한 다른 시약들의 양도 정확히 준수해야 정확한 PCR 결과가 나올 것이다.
다음으로 PCR 과정에서의 오염이 오차의 원인이라고 생각한다. PCR 과정에서의 오염은 증폭되기 전 DNA가 공기 입자 등 다양한 접촉 경로로 인해 오염되는 Cross-contamination과 PCR을 마친 뒤 증폭된 DNA가 그 다음 PCR을 진행하는 과정에서 오염되는 Carryover contamination가 있다. 이번 실험의 경우 PCR을 진행하기 전 오염되었을 가능성이 크기 때문에 Cross-contamination이 발생했을 수 있다고 생각한다. 따라서 오염을 방지하기 위해서 사용하는 PCR tube와 피펫 팁, 용액 등을 멸균해 핵산 분해효소를 불활성 시켜두고, 실험 과정에서 실험복과 실험 장갑을 반드시 착용해야 실험의 정확도를 높일 수 있다.
5. Reference
[1] pET-22b(+) vector Protocol
[2] 미생물 실험서/ 유민, 김병오, 이혜영 공저/ p.68-75
(4) 전기영동 기기에 Agarose gel을 넣고 1X TAE buffer를 잠길 때까지 채운다.
(5) 1kbp DNA ladder와 6X Loading star를 섞어 Agarose gel의 첫 번째 홈에 loading 한다.
(6) PCR 샘플과 6X Loading star를 섞어 두 번째 홈에 loading 한다.
(7) 100 V의 전압에서 1시간 동안 전기영동을 실시한다.
(8) Agarose gel을 꺼내 UV transilluminator 위에 놓고 UV를 쬐어 PCR의 결과를 확인한다.
※ 실험 시 유의사항
- 의 온도조절에 유의한다. 가 너무 높으면 프라이머가 주형 DNA에 너무 약하게 결합되어 PCR산물의 양이 너무 적고, 가 너무 낮으면 프라이머가 비특이적으로 결합해 원하지 않는 DNA가 증폭될 수 있기 때문이다.
- 핵산 분해효소는 손이나 땀 등에도 있기 때문에 혼입을 막기 위해 장갑과 마스크를 반드시 착용해야 한다.
- 사용하는 tube와 피펫 팁, 용액 등은 멸균해 핵산 분해효소를 불활성 시켜둬야 한다.
3. Result
PCR의 결과로 증폭된 Target DNA의 전기영동 결과는 다음과 같다.
Fig 13. 전기영동 결과
이번 실험에서 모든 조의 전기영동 결과가 대체로 희미하게 나왔고, 1조와 4조와 6조가 조금 모양이 보이며 그 중 1조가 가장 잘 보이는 상태이다. 희미한 부분을 첫 번째 홈에 loading한 DNA ladder (기준이 되는 샘플)와 우리 6조의 전기영동 결과를 비교해보면, 2.0kbp과 3.0kbp 사이에 위치하며 3.0kbp쪽에 더 가까우므로 대략 2.8kb라고 측정할 수 있다. 하지만 결과가 매우 희미해 정확한 확인이 쉽지 않아 조교님께서 추가적으로 정상상태의 밴드사진을 올려주셨다. 이를 통해 정상상태의 Target DNA의 크기를 측정해보자.
Fig 14. 조교님께서 첨부해주신 정상상태의 전기영동 결과
Fig 14.를 살펴보면, 정상상태의 밴드 또한 이번 실험 결과와 마찬가지로 Target DNA의 크기가 대략 2.8kbp라는 것을 알 수 있다.
4. Discussion
전기영동 결과 우리 조의 밴드 중 가장 밝은 부분의 크기를 측정하니 대략 2.8kbp였다. 하지만 우리 조를 포함한 모든 조의 밴드가 대체적으로 너무 흐릿하고 희미하게 나와 정확한 판별이 힘들었다. 조교님이 첨부해주신 정상상태 밴드에서의 크기를 측정하니 마찬가지로 2.8kbp로 측정되었다. 이렇게 전기영동의 결과 Target DNA의 크기가 2.8kbp인 이유와 전기영동 결과가 희미하게 제대로 나오지 않은 이유에 대해 분석해보자.
1) 전기영동의 결과 Target DNA의 크기가 2.8kbp인 이유
이번 실험에 사용한 plasmid DNA는 유전자 재조합된 pET-22b(+)이며, PCR mixture 제작에 사용한 Forward primer는 T7 primer(T7 promoter), Reverse primer는 T7 Terminator primer이다. pET-22b(+)의 염기 서열 중 이 두 프라이머의 sequence landmarks는 다음과 같다.
Fig 15. T7 promoter와 T7 Terminator의 sequence landmarks
따라서 T7 promotor는 361-377, T7 terminator는 26-72이고, Forward primer와 Reverse primer는 T7 promoter와 T7 Terminator의 부위에 상보적으로 결합해 DNA의 합성을 진행하므로 우리가 기대할 수 있는 Target DNA의 크기는 (377-26= 351bp =0.351kbp)이다. 그러나 eGFP (0.8kpb)와 another gene (1.7kbp) 또한 함께 증폭되었을 것이다. 따라서 우리가 이번에 사용한 복제할 DNA의 크기인 2.5kbp에 0.351kbp의 길이를 더해주면 이번 실험에서 얻을 수 있는 이론적인 Target DNA의 크기는 2.851kbp가 나오게 된다.
2) 전기영동 결과가 희미하게 제대로 나오지 않은 이유
먼저 가장 큰 오차의 원인은 PCR mixture를 만드는 과정에서 발생했다고 생각한다. PCR mixture를 만들기 위해 넣은 시약 중 Reverse primer (T7 terminator primer)는 넣어야 하는 양이 0.5 μl로 극도로 적은 양이었기 때문에 가장 적은 용량의 마이크로 피펫과 피펫 팁을 사용해 정확히 넣어보려 했지만 거의 눈에 보이지 않을 정도로 작아 용액이 들어갔는지 확인하는 것조차 쉽지 않았다. 이러한 정확하게 넣지 못한 용액의 양으로 인해 오차가 발생했을 수 있다고 생각한다. Reverse primer는 Target DNA의 증폭에 있어 그 범위를 정해주는 매우 중요한 시약이기 때문에 정량의 오차가 PCR 과정에서의 오류를 유발시킬 수 있다. 또한 다른 시약들의 양도 정확히 준수해야 정확한 PCR 결과가 나올 것이다.
다음으로 PCR 과정에서의 오염이 오차의 원인이라고 생각한다. PCR 과정에서의 오염은 증폭되기 전 DNA가 공기 입자 등 다양한 접촉 경로로 인해 오염되는 Cross-contamination과 PCR을 마친 뒤 증폭된 DNA가 그 다음 PCR을 진행하는 과정에서 오염되는 Carryover contamination가 있다. 이번 실험의 경우 PCR을 진행하기 전 오염되었을 가능성이 크기 때문에 Cross-contamination이 발생했을 수 있다고 생각한다. 따라서 오염을 방지하기 위해서 사용하는 PCR tube와 피펫 팁, 용액 등을 멸균해 핵산 분해효소를 불활성 시켜두고, 실험 과정에서 실험복과 실험 장갑을 반드시 착용해야 실험의 정확도를 높일 수 있다.
5. Reference
[1] pET-22b(+) vector Protocol
[2] 미생물 실험서/ 유민, 김병오, 이혜영 공저/ p.68-75






























소개글