목차
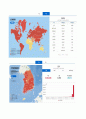
1. COVID-19 관련 데이터 시각화의 국내외 사례 3개를 찾고 비교하여 정리하시오. (반드시 2022년 데이터가 포함된 사례를 사용할 것. 이미지를 캡처하여 한글이나 워드 파일에 첨부할 것. 이미지를 별도의 파일로 제출하지 말 것) (6점)
2. 한스 로즈링의 TED 강의(아래의 URL 이용)를 보고 데이터 시각화의 역할 등 느낀 점을 1페이지 이내로 정리하시오.(6점) https://www.ted.com/talks/hans_rosling_let_my_dataset_change_your_mindset (오른쪽 아래 메뉴에서 한글 자막 설정 가능)
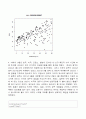
3. R의 datarium 패키지에 내장된 marketing 데이터셋은 광고 미디어에 사용한 비용과 판매액의 데이터이다. facebook 컬럼은 facebook 광고비로 사용한 금액이고, sales 컬럼은 판매액이다. facebook을 x축, sales를 y축으로 하는 산점도를 그리시오. facebook을 독립변수(설명변수), sales를 종속변수(반응변수, 결과변수)로 하는 회귀직선을 산점도 위에 그리시오. 산점도의 제목으로 본인의 학번을 출력하시오. (9점) (힌트: datarium 패키지를 설치, 로드한 후 콘솔에 dat<-marketing을 입력하면 marketing 데이터셋이 dat에 저장된다)
4. 한국, 미국, 프랑스, 일본의 COVID-19 신규 확진자 수의 시간에 따른 추이를 데이터 시각화로 비교하고 향후 추이에 대해 의견 기술하라(데이터는 과제 작성일까지 올라와 있는 것을 이용하면 되며 학생별로 동일할 필요는 없음).(9점)
*데이터 소스: https://ourworldindata.org/covid-deaths 에서 “Our work belongs to everyone“이라 쓰여있는 네모 안의 .csv(아래 그림에서 빨간 네모)를 클릭하여 데이터를 다운로드 받아서 사용할 것.
*변수이름: iso_code가 각 국가를 나타낸다. (한국: KOR, 미국:USA, 프랑스: FRA, 일본: JPN)
new_cases가 신규 확진자 수를 나타낸다.
2. 한스 로즈링의 TED 강의(아래의 URL 이용)를 보고 데이터 시각화의 역할 등 느낀 점을 1페이지 이내로 정리하시오.(6점) https://www.ted.com/talks/hans_rosling_let_my_dataset_change_your_mindset (오른쪽 아래 메뉴에서 한글 자막 설정 가능)
3. R의 datarium 패키지에 내장된 marketing 데이터셋은 광고 미디어에 사용한 비용과 판매액의 데이터이다. facebook 컬럼은 facebook 광고비로 사용한 금액이고, sales 컬럼은 판매액이다. facebook을 x축, sales를 y축으로 하는 산점도를 그리시오. facebook을 독립변수(설명변수), sales를 종속변수(반응변수, 결과변수)로 하는 회귀직선을 산점도 위에 그리시오. 산점도의 제목으로 본인의 학번을 출력하시오. (9점) (힌트: datarium 패키지를 설치, 로드한 후 콘솔에 dat<-marketing을 입력하면 marketing 데이터셋이 dat에 저장된다)
4. 한국, 미국, 프랑스, 일본의 COVID-19 신규 확진자 수의 시간에 따른 추이를 데이터 시각화로 비교하고 향후 추이에 대해 의견 기술하라(데이터는 과제 작성일까지 올라와 있는 것을 이용하면 되며 학생별로 동일할 필요는 없음).(9점)
*데이터 소스: https://ourworldindata.org/covid-deaths 에서 “Our work belongs to everyone“이라 쓰여있는 네모 안의 .csv(아래 그림에서 빨간 네모)를 클릭하여 데이터를 다운로드 받아서 사용할 것.
*변수이름: iso_code가 각 국가를 나타낸다. (한국: KOR, 미국:USA, 프랑스: FRA, 일본: JPN)
new_cases가 신규 확진자 수를 나타낸다.
본문내용
pch=19)
title(\" 학번 : 202028-352807\")
sale.lm=lm(y ~ x, data=dat2)
abline(sale.lm)
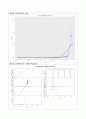
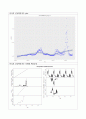
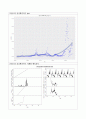
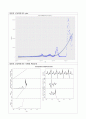
4. 아래의 그림은 한국, 미국, 프랑스, 일본의 COVID-19 신규 확진자 수의 시간에 따른 추이를 나타내고 있고 추가적인 타임테이블을 통한 분석을 하였다. 한국의 경우는 2022년 2월 이후 신규확진자가 폭발적으로 증가하였다. 그러나 미국의 경우는 2022년 말과 2021년초에 대유행이 있었고 2021년 9월 경에 또 한번의 대유행이후 2022년 1월 정점을 기준으로 감소하고 있다. 프랑스의 경우도 미국과 유사하게 움직이고 있으며 일본의 경우는 2021년 9월경 대유행이후 2022년 2월과 3월 폭증하였고 지금은 감소세로 보인다. 미국의 경우는 정점을 지났으며 한국, 프랑스, 일본은 이제 탈출구 쪽에 와 있는 것으로 보인다. 미국의 경우가 오히려 안정국면을 더 빨리 찾았고 탈출구를 벗어 났다고 보인다. 앞으로는 모든 나라에서 감소세가 될 것으로 전망할수 있다. 특히 호흡기 질병으로 인하여 프랑스, 일본, 미국의 경우 계절별로 2차례나 3차례의 폭증이 있었음을 알수 있다. 만일 코로나가 안정된다고 하여도 계절적인 유행은 또 다시 일시적으로 올수 있고 이러한 시계열 자료를 통하여 반복적인 패턴을 보일것으로 예측된다.
install.packages(\"readxl\")
setwd(\"C:/data\")
library(readxl)
install.packages(\"TTR\")
library(TTR)
install.packages(\"forecast\")
library(forecast)
install.packages(\"tseries\")
library(tseries)
install.packages(\"ggplot2\")
library(ggplot2)
covid=read_excel(\"covid.xlsx\")
kor_covid = subset(covid, covid$iso_code == \"KOR\")
kor_covid1 = kor_covid[,4:6]
kor_covid2 = kor_covid1[,-2]
p = ggplot(data=kor_covid2, aes(x=date, y=new_cases)) + geom_point(shape=15, size=1, colour=\"blue\") + geom_smooth()
p +labs(title=\"한국의 COVID-19 신규 확진자 수\",
x =\"시간\", y = \"신규확진자\") +
theme(plot.title = element_text(hjust = 0.5))
kor_covid3 = ts(kor_covid2, frequency = 365, start = c(2020,2,24))
kor_covid3_comp <- decompose(kor_covid3)
plot(kor_covid3_comp)
# 일본 분석
jpn_covid = subset(covid, covid$iso_code == \"JPN\")
jpn_covid1 = jpn_covid[,4:6]
jpn_covid2 = jpn_covid1[,-2]
p = ggplot(data=jpn_covid2, aes(x=date, y=new_cases)) + geom_point(shape=15, size=1, colour=\"blue\") + geom_smooth()
p +labs(title=\"일본의 COVID-19 신규 확진자 수\",
x =\"시간\", y = \"신규확진자\") +
theme(plot.title = element_text(hjust = 0.5))
jpn_covid3 = ts(jpn_covid2, frequency = 365, start = c(2020,2,24))
jpn_covid3_comp <- decompose(jpn_covid3)
plot(jpn_covid3_comp)
# 미국 분석
usa_covid = subset(covid, covid$iso_code == \"USA\")
usa_covid1 = usa_covid[,4:6]
usa_covid2 = usa_covid1[,-2]
p = ggplot(data=usa_covid2, aes(x=date, y=new_cases)) + geom_point(shape=15, size=1, colour=\"blue\") + geom_smooth()
p +labs(title=\"미국의 COVID-19 신규 확진자 수\",
x =\"시간\", y = \"신규확진자\") +
theme(plot.title = element_text(hjust = 0.5))
usa_covid3 = ts(usa_covid2, frequency = 365, start = c(2020,2,24))
usa_covid3_comp <- decompose(usa_covid3)
plot(usa_covid3_comp)
# 프랑스 분석
fra_covid = subset(covid, covid$iso_code == \"FRA\")
fra_covid1 = fra_covid[,4:6]
fra_covid2 = fra_covid1[,-2]
p = ggplot(data=fra_covid2, aes(x=date, y=new_cases)) + geom_point(shape=15, size=1, colour=\"blue\") + geom_smooth()
p +labs(title=\"프랑스의 COVID-19 신규 확진자 수\",
x =\"시간\", y = \"신규확진자\") +
theme(plot.title = element_text(hjust = 0.5))
df_fra_covid2 <- na.omit(fra_covid2)
fra_covid3 = ts(df_fra_covid2, frequency = 365, start = c(2020,2,24))
fra_covid3_comp <- decompose(fra_covid3)
plot(fra_covid3_comp)
한국의 신규확진자수 plot
한국의 신규확진자수 시계열 예측분석
미국의 신규확진자수 plot
미국의 신규확진자수 시계열 예측분석
프랑스의 신규확진자수 plot
프랑스의 신규확진자수 시계열 예측분석
일본의 신규확진자수 plot
일본의 신규확진자수 시계열 예측분석
title(\" 학번 : 202028-352807\")
sale.lm=lm(y ~ x, data=dat2)
abline(sale.lm)
4. 아래의 그림은 한국, 미국, 프랑스, 일본의 COVID-19 신규 확진자 수의 시간에 따른 추이를 나타내고 있고 추가적인 타임테이블을 통한 분석을 하였다. 한국의 경우는 2022년 2월 이후 신규확진자가 폭발적으로 증가하였다. 그러나 미국의 경우는 2022년 말과 2021년초에 대유행이 있었고 2021년 9월 경에 또 한번의 대유행이후 2022년 1월 정점을 기준으로 감소하고 있다. 프랑스의 경우도 미국과 유사하게 움직이고 있으며 일본의 경우는 2021년 9월경 대유행이후 2022년 2월과 3월 폭증하였고 지금은 감소세로 보인다. 미국의 경우는 정점을 지났으며 한국, 프랑스, 일본은 이제 탈출구 쪽에 와 있는 것으로 보인다. 미국의 경우가 오히려 안정국면을 더 빨리 찾았고 탈출구를 벗어 났다고 보인다. 앞으로는 모든 나라에서 감소세가 될 것으로 전망할수 있다. 특히 호흡기 질병으로 인하여 프랑스, 일본, 미국의 경우 계절별로 2차례나 3차례의 폭증이 있었음을 알수 있다. 만일 코로나가 안정된다고 하여도 계절적인 유행은 또 다시 일시적으로 올수 있고 이러한 시계열 자료를 통하여 반복적인 패턴을 보일것으로 예측된다.
install.packages(\"readxl\")
setwd(\"C:/data\")
library(readxl)
install.packages(\"TTR\")
library(TTR)
install.packages(\"forecast\")
library(forecast)
install.packages(\"tseries\")
library(tseries)
install.packages(\"ggplot2\")
library(ggplot2)
covid=read_excel(\"covid.xlsx\")
kor_covid = subset(covid, covid$iso_code == \"KOR\")
kor_covid1 = kor_covid[,4:6]
kor_covid2 = kor_covid1[,-2]
p = ggplot(data=kor_covid2, aes(x=date, y=new_cases)) + geom_point(shape=15, size=1, colour=\"blue\") + geom_smooth()
p +labs(title=\"한국의 COVID-19 신규 확진자 수\",
x =\"시간\", y = \"신규확진자\") +
theme(plot.title = element_text(hjust = 0.5))
kor_covid3 = ts(kor_covid2, frequency = 365, start = c(2020,2,24))
kor_covid3_comp <- decompose(kor_covid3)
plot(kor_covid3_comp)
# 일본 분석
jpn_covid = subset(covid, covid$iso_code == \"JPN\")
jpn_covid1 = jpn_covid[,4:6]
jpn_covid2 = jpn_covid1[,-2]
p = ggplot(data=jpn_covid2, aes(x=date, y=new_cases)) + geom_point(shape=15, size=1, colour=\"blue\") + geom_smooth()
p +labs(title=\"일본의 COVID-19 신규 확진자 수\",
x =\"시간\", y = \"신규확진자\") +
theme(plot.title = element_text(hjust = 0.5))
jpn_covid3 = ts(jpn_covid2, frequency = 365, start = c(2020,2,24))
jpn_covid3_comp <- decompose(jpn_covid3)
plot(jpn_covid3_comp)
# 미국 분석
usa_covid = subset(covid, covid$iso_code == \"USA\")
usa_covid1 = usa_covid[,4:6]
usa_covid2 = usa_covid1[,-2]
p = ggplot(data=usa_covid2, aes(x=date, y=new_cases)) + geom_point(shape=15, size=1, colour=\"blue\") + geom_smooth()
p +labs(title=\"미국의 COVID-19 신규 확진자 수\",
x =\"시간\", y = \"신규확진자\") +
theme(plot.title = element_text(hjust = 0.5))
usa_covid3 = ts(usa_covid2, frequency = 365, start = c(2020,2,24))
usa_covid3_comp <- decompose(usa_covid3)
plot(usa_covid3_comp)
# 프랑스 분석
fra_covid = subset(covid, covid$iso_code == \"FRA\")
fra_covid1 = fra_covid[,4:6]
fra_covid2 = fra_covid1[,-2]
p = ggplot(data=fra_covid2, aes(x=date, y=new_cases)) + geom_point(shape=15, size=1, colour=\"blue\") + geom_smooth()
p +labs(title=\"프랑스의 COVID-19 신규 확진자 수\",
x =\"시간\", y = \"신규확진자\") +
theme(plot.title = element_text(hjust = 0.5))
df_fra_covid2 <- na.omit(fra_covid2)
fra_covid3 = ts(df_fra_covid2, frequency = 365, start = c(2020,2,24))
fra_covid3_comp <- decompose(fra_covid3)
plot(fra_covid3_comp)
한국의 신규확진자수 plot
한국의 신규확진자수 시계열 예측분석
미국의 신규확진자수 plot
미국의 신규확진자수 시계열 예측분석
프랑스의 신규확진자수 plot
프랑스의 신규확진자수 시계열 예측분석
일본의 신규확진자수 plot
일본의 신규확진자수 시계열 예측분석
추천자료
 (출석대체 관광문화교양4) 세계의도시와건축 - 파시브하우스의 장점과 단점을 정리
(출석대체 관광문화교양4) 세계의도시와건축 - 파시브하우스의 장점과 단점을 정리 (출석대체 유아교육3) 유아언어교육 - 유아의 혼잣말에 대해 피아제와 비고츠키의 주장을 비교
(출석대체 유아교육3) 유아언어교육 - 유아의 혼잣말에 대해 피아제와 비고츠키의 주장을 비교 (출석대체 청소년2) 청소년상담 - 상담 기법에 대해 설명한 후, 이 중 2가지 기법을 일상 속...
(출석대체 청소년2) 청소년상담 - 상담 기법에 대해 설명한 후, 이 중 2가지 기법을 일상 속... (기말 교양1) 인터넷과정보사회 - 메타버스, 일하는 방식을 바꾸다
(기말 교양1) 인터넷과정보사회 - 메타버스, 일하는 방식을 바꾸다 (기말 문화관광4) 세계의도시와건축 - 런던의 19세기 콜레라 유행과 21세기 런던의 코로나바...
(기말 문화관광4) 세계의도시와건축 - 런던의 19세기 콜레라 유행과 21세기 런던의 코로나바... (기말 문화관광4) 세계의도시와건축 - 함부르크의 하수 시설과 서울의 하수 시설 변천
(기말 문화관광4) 세계의도시와건축 - 함부르크의 하수 시설과 서울의 하수 시설 변천 (기말 미디어3) 현대광고와카피전략 - 광고의 커뮤니케이션 기능으로 3가지 있는데 이 3가지 ...
(기말 미디어3) 현대광고와카피전략 - 광고의 커뮤니케이션 기능으로 3가지 있는데 이 3가지 ... (2022년 방송통신대 데이터시각화 기말과제물)런던 콜레라지도를 조사하고 데이터 시각화의 ...
(2022년 방송통신대 데이터시각화 기말과제물)런던 콜레라지도를 조사하고 데이터 시각화의 ...



























소개글